For a brief introduction to our lab, see the following brochure and flyer: Brochure2019
We decode bio-complexity to unravel/control the hidden logic of life and to apply it for novel drug discovery and precision medicine.
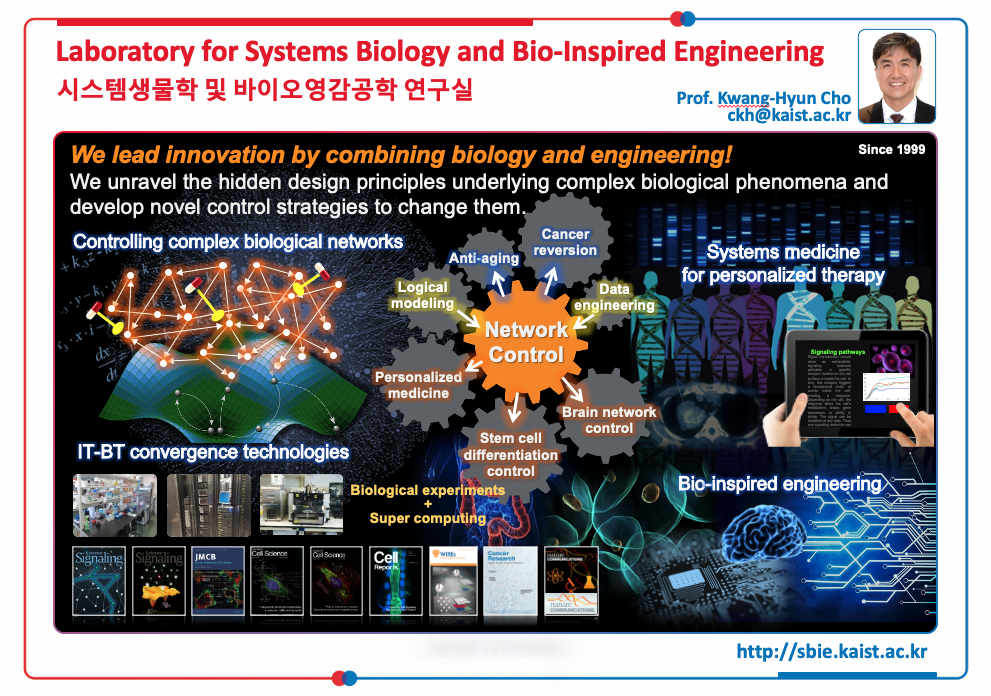
The Life Sciences are witnessing a shift of paradigm from traditional characterization of individual molecules towards an understanding of interactive pathways and networks. The role of genes, proteins, metabolites and cells can be understood and defined through their interactions and it is through our focus on intra- and inter-cellular dynamics that we are deeply involved in the emerging area of Systems Biology. For Systems Biology to succeed, we have to cope with the bewildering complexity of cellular systems, covering a wide range of spatial and temporal scales. Two of the key characteristics of Systems Biology are dynamic modeling and integration (fusion) of various information sources, such as genomics, transcriptomics, proteomics, and metabolomics. In this context, our research has been centered on systems-level investigations of cellular signal transduction pathways, reverse engineering of biomolecular regulatory networks, and the unraveling of hidden cellular dynamics. We focus on developing a systems biology analysis of cellular information processing by signaling and gene networks in cells, with particular emphasis towards understanding cell-fate decisions on proliferation and differentiation. Regulation of the commitment to differentiation is central to many biological processes such as cancer, inflammatory diseases, and neurodegeneration. Our research is driven by two long term objectives: (1) to create a predictive model for a reprogrammable cell that can be optimized and controlled for personalized therapy and drug discovery, and (2) to apply the knowledge obtained from the study of biological systems to engineering. In this way we hope to contribute to engineering innovation using ideas inspired by molecular systems biology.
What is Systems Biology?
Systems biology refers to the quantitative analysis of the dynamic interactions among multiple components of a biological system and aims to understand/control the behavior of the system as a whole. Systems biology involves the development and application of system-theoretic concepts for the study of complex biological systems through iteration over mathematical modeling, computer simulations and biological experiments. Systems biology can be viewed as new concept and tool to increase our understanding of the working/design principle of biological systems, to develop more directed experiments, to allow accurate predictions, and eventually to control the dynamic behavior of a biological system.
Systems biology is among the most rapidly growing fields of scientific research and technology development. Over the decades, the research activities in this field have been increasing exponentially. This is evidenced by the number of published papers as well as the growing number of conferences, journals, research institutions and centers, and academic events on the topic of systems biology. Systems biology is a newly emerged, continuously evolving, interdisciplinary field of study on living systems driven by unusual curiosity.
Recent advances in single-cell omics measurement technologies have ushered in a new renaissance in systems biology. In particular, the ability to measure diverse omics data at the single-cell level simultaneously, and extract time-dependent information through pseudo-time ordering, as if conducting a time-series experiment, has revolutionized the field. This approach allows for the construction of more precise intracellular molecular network models based on data that previously required enormous effort to measure. As a result, key mechanisms of cellular fate changes can now be pinpointed with single-cell precision, and even manipulated in desired directions. Single-cell systems biology, driven by single-cell multi-omics data, holds the potential to unlock answers to fundamental questions about the origins of life, as well as provide new therapeutic strategies for treating intractable diseases. Our laboratory is particularly focused on solving the mysteries of cancer and aging, with the goal of finding novel ways to reverse these processes.
For further reading: W. Dubitzky, O. Wolkenhauer, K.-H. Cho, and H. Yokota (Editors-in-Chief), Encyclopedia of Systems Biology, Springer, New York, Vol. 1-4, 2013.
시스템생물학이란?
“세포의 모든 구성 분자들을 규명하면 우리는 과연 인공세포를 만들 수 있을까?”
지금까지 생명과학에서는 생명현상에 관여하는 주요 물질을 발견하고 그 분자적, 기능적 특징을 규명하는데 초점을 맞추어왔을 뿐 이들이 어떻게 상호작용하며 생명‘시스템’을 구성하는지에 대해서는 그다지 관심을 기울이지 않았다. 그러나 생명의 본질을 이해하고 복잡한 생명현상을 지배하고 있는 근본적인 원리를 밝혀내려면 생명시스템을 이루는 성분들간의 구성과 동역학(dynamics)적 특성을 집합적으로 이해해야 한다. 이를 위해서는 모든 생체현상의 원인과 결과를 초래하는 분자, 세포, 조직, 장기, 그리고 유기체들 각각에 있어서 시공간적 상호관계를 연구해야한다. 생명의 가장 기본단위인 단일 세포의 작동원리를 이해하는데 있어서 문제의 핵심은 세포의 작용이 다수의 유전자, 단백질 및 다른 분자들간의 복잡한 상호작용에 의해 조절된다는 것이다. 시스템생물학의 궁극적인 목적은 이러한 조절작용의 본질을 이해함으로써 생명현상 속에 잠재된 지배원리에 대해 보다 깊은 통찰력을 얻고 나아가 생명현상을 인위적으로 조절해보고자 하는 것이다. 이러한 목표는 전통적인 생물학 연구방식이 추구해온 생명시스템의 구성요소 발견과 발견된 요소의 물리/화학적 특징을 규명하는 것만으로는 결코 이루어질 수 없으며 수학 모델링을 통한 정보의 재구성과 이를 토대로 분자단위 혹은 그 이상의 레벨에서 이루어지는 구성요소들간의 네트워크와 신호전달경로 등을 시스템 차원에서 분석, 시뮬레이션 해봄으로써 가능하다. 최근 연이은 기술의 진보로 인해 생명현상에 대한 보다 정밀하고 다양한 측정이 가능해짐에 따라 측정된 데이터를 시스템 차원에서 재해석하기 위해 필요한 고유의 이론과 방법론 개발이 시스템생물학이라는 신기술 융합연구로서 새로운 패러다임을 형성하며 급부상하고 있다. 시스템생물학은 시스템이론을 생명과학에 응용하여 생체구성요소들의 상호관계와 상호작용을 분석, 규명함으로써 생명현상에 대한 시스템 차원의 이해를 도모하는 학제간 신기술 융합분야이다. 시스템생물학은 생체구성요소들이 개별적으로 존재할 때에는 보이지 않는, 즉 상호작용에 의해 새롭게 표출되는 (소위 ‘창발적’인) 생명의 기능을 탐구하는데 그 초점이 있다.
생명체의 DNA 염기서열 분석 및 유전자 해독과 더불어 유전체학, 분자생물학, 생화학, 물리생물학 등의 발전으로 인해 생명시스템의 구성세포에 대한 분자수준의 이해가 가능해졌으며 이로 인해 더 많은 정보를 수집할 수 있는 도구의 개발이 이루어지게 되었다. 생명시스템의 구성세포들이 복잡한 동역학 특성을 지닌 정교한 제어시스템이라는 사실은 이미 오래 전부터, 특히 실험생물학자들에 의해, 잘 인지된 사실이지만 오늘날까지 실제로 그러한 개념을 바탕으로 실험이 계획되거나 실험결과가 분석되지는 않고 있다. 즉, 분자생물학에서 사용되고 있는 많은 실험기법들은 주로 유전자 또는 단백질에 대한 분자적, 기능적 특징을 기술하는데 초점이 맞추어져 있다. 그러나 생명시스템을 구성하는 세포의 기능을 결정짓는 동역학 특성을 분석하기 위해서는 어떠한 표현형에 관련된 개별 유전자나 단백질을 식별해내려는 시도보다 관측결과를 세포내부의 동역학에 의한 일련의 상태변화로 간주하고 이를 탐구하는 시도가 필요하다. 시스템생물학의 출발은 이렇듯 어떤 표현형의 원인 분자를 찾기보다 그러한 결과를 초래한 세포내부의 동역학 특성을 규명하려는 시도에서 비롯된다. 즉, 생명시스템의 기능과 오작동 등 생체기능을 이해함에 있어서 최대 난제는 생명시스템 구성요소들의 복잡한 상호작용을 밝혀내는 것인데 이를 위해서는 전통적인 분자생물학적 접근보다 시스템생물학적 접근이 더욱 중요한 열쇠를 지니고 있다.
시스템생물학의 어원자체는 시스템과학(systems science)과 생물학(biology)의 합성어이다. 여기에서 ‘시스템’의 아이디어를 이해하기 위해 제어공학(control engineering)을 비롯한 기존 시스템과학에서 다루어온 시스템이론(systems theory)을 살펴보면 지금까지 공학이라는 전혀 다른 영역 속에서 오랜 역사를 지니며 발전해왔음을 알 수 있다. 이러한 시스템이론을 개념적으로 요약하면 단순한 기술적 지식의 총체라기 보다는 어떠한 물리적 현상 또는 그 대상체의 상태변수들간 상호관계 및 동역학 특성 등을 규명하고 이를 바탕으로 외부의 인위적 조작을 통해 원하는 출력, 성능 등을 획득함으로써 분석 및 예측 가능한 모델을 정립할 수 있도록 하는 체계적 사고방식이라 할 수 있다. 시스템생물학을 통한 시스템과학과 생명과학의 자연스러운 결합은 이미 역사적으로 오래 전에 언급되었으며 여러 선지적 과학자들이 관련 연구를 시도한 바 있다. 시스템생물학의 기원은 1929년 캐논(Cannon)의 항상성(homeostasis) 개념과 유기체가 동적제어시스템이라고 언급한 사실로부터 찾을 수 있다. 이후 1945년 버터란피(Bertalanffy)는 유기체를 개방형시스템(open system) 이론으로 기술하였으며, 위너(Wiener)는 동물과 기계에서 공존하는 제어와 통신의 개념에 대해 기술함으로서 사이버네틱스(cybernetics)의 효시를 마련하였다. 1958년 애쉬비(Ashby)는 적응, 자기구성 능력을 가진 유기체를 기계에 비유하였고, 한편 로젠(Rosen)은 엠알(M,R)이론을 통해 유기체와 기계의 상이성을 이론적으로 승화시켰다. 시스템생물학이라는 용어는 1968년 메사로비치(Mesarovic)에 의해 최초로 사용되었는데 당시 시스템생물학 연구의 성공을 위해 지적한 두 가지 사항, 즉 시스템과학자들(제어공학자, 수학자, 물리학자)은 생명과학의 의미있는 문제해결을 위해 실험생물학자들과 밀접한 공동연구를 수행해야 하며, 또한 생물학자들은 시스템과 신호, 그리고 동적 상호작용의 관점에서 생명현상을 바라보아야 한다고 한 것은 현 시점에 비추어 보아도 매우 의미심장하다. 1970년 자콥(Jacob)과 모노드(Monod)는 조절단백질의 작용으로부터 세포 사이버네틱스(cell cybernetics)를 고안하였으며, 1975년 세겔(Segel)은 효소역학(enzyme kinetics)을 정립하였고, 1978년 밀러(Miller)는 시스템이론을 생명현상으로부터 국가전체의 사회현상에 이르기까지 적용하여 총체적 메커니즘을 해석하려는 시도를 하였다. 1996년 하인리히(Heinrich)와 슈스터(Schuster)는 생체 세포시스템의 조절작용에 관해 기술하였고, 골드버터(Goldbeter)는 생화학적 진동현상 및 세포주기에 관해 연구하였으며, 1997년 펠(Fell)은 대사과정의 제어메커니즘에 관해 분석 기술하였다. 현대 생명과학계에서는 벤터(Venter)(1999), 하트웰(Hartwell)(1999), 널스(Nurse)(2000), 프레이저(Fraser)(2000) 등의 여러 과학자들이 <네이처>와 <셀> 등의 저널을 통해 생명현상을 기술할 수 있는 일반적인 시스템이론의 필요성과 이를 통한 체계적이고 정량적인 현상의 예측에 관해 언급함으로써 시스템생물학의 필연적인 시대적 재조명을 예견하기에 이르렀다. 이러한 역사적 배경가운데 위버(Weaver)(1948), 마인하트(Meinhardt)(1988), 카우프만(Kauffman)(1995), 헤이켄(Haken)(1997), 해리슨(Harrison)(1993), 굿윈(Goodwin)(2001) 등에 의한 자연계의 비조직적 복잡성(disorganized complexity)과 조직적 복잡성(organized complexity)에 대한 연구는 현재의 시스템생물학 연구에 직접적 동기를 제공하였다. 즉, 시스템생물학은 이러한 복잡계에 대한 연구를 현대 생명과학의 포스트 게놈시대로 연장하며 재조명받게 된 분야라고 할 수 있다. 그러나 시스템생물학은 생명시스템의 동역학 특성을 신호 및 시스템적 접근법을 통해 기술한다는 측면에서 복잡계의 연구와 분명 차별화 된다.
최근 기술의 진보로 인해 가능해진 대량의 분자생물학 실험데이터의 생성은 데이터의 효율적 관리와 분석을 위해 자연스럽게 컴퓨터과학과 통계학의 도움을 필요로 하게 되었으며 이로 인해 생명과학의 발전은 생물정보학(bioinformatics)이라는 신분야의 창출과 발전으로 이어지게 되었다. 그러나 게놈 서열데이터가 이제 이용가능해짐에 따라 서열분석이나 분자적 특징을 규명하는 생물정보학의 범주로부터 더 나아가 유전자, 단백질 등의 기능과 상호작용을 이해하려는 시스템생물학으로 관심의 초점이 옮겨지고 있다. 시스템생물학의 보다 근원적인 목표는 단순히 생명현상에서 관측되는 상관관계(correlation)를 분석하는 것이 아니라 생명시스템이 외부 자극에 대해 어떻게 내부적인 동역학 변화를 유발하고 이로 인해 자극에 대한 반응을 비로소 표출하는지에 대한 인과관계(causality)를 밝히고자 하는 것이다. 생명시스템 구성의 기본 단위인 세포는 시공간상에서 상호작용하는 분자들의 ‘네트워크(network)’로 이루어져 있으며 자가조절기능을 갖춘 일종의 동역학시스템이다. 그런데 이러한 세포 네트워크의 구조, 기능, 그리고 조절작용 등을 지배하는 상호관계는 현재까지 대부분 밝혀져 있지 않다. 시스템생물학은 실험과 이론적 방법론 개발의 융합연구를 통해 이러한 상호관계를 규명하고 설명하려는데 그 목적이 있다. 이렇듯 시스템생물학 연구가 새롭게 중요시되는 이유는 종래의 생물정보학에서 데이터를 마이닝(mining)하는 방식으로는 구성요소 혹은 변수들간의 상관관계만을 밝힐 수 있을 뿐 정작 필요한 상호관계와 상호작용 등에는 접근할 수 없기 때문이다. 즉, 더 이상 세포의 분자적 특성을 볼트와 너트의 조합만으로 이해하기 보다 그 본질적 기능을 탐구하기 위해서 게놈데이터의 마이닝 방식이 아닌 시스템과 신호 관점에서의 방법론 개발이 절실하다는 데에 초점이 집중되고 있다. 시스템생물학은 보다 많은 실험적 발견과 지식을 집대성하기 보다 새로운 ‘시스템’적 사고로의 전환을 요한다.
시스템생물학이 수십년간 역사속으로 사라졌다가 근래에 이르러 재조명을 받게 된 이유 가운데 하나는 측정기술의 진보로 인해 시스템이론의 적용이 가능한 수준의 정량적 데이터를 얻을 수 있게 된 사실에 있다. 그러한 예로서 다양한 형태의 유전자칩(DNA microarray, RNAseq, Nanostring 등) 기술을 들 수 있다. 이를 통해 동시에 여러 유전자의 발현정도를 비교 분석할 수 있게 되었기 때문이다. 그러나 이 경우 생물정보학의 수준을 넘어서 시스템생물학 연구를 수행하기 위해서는 동역학 특성을 분석할 수 있는 시계열 데이터의 생성이 필요하며 여기에는 한편으론 아직 데이터의 품질향상, 정보의 불확실성 제거, 샘플링 숫자에 비해 상대적으로 많은 변수개수의 처리문제 등 또다시 선결되어야 할 많은 숙제들이 남아있다. 그러나 데이터의 질적 수준이 꾸준히 향상되고 있으며 실험비용 또한 점차 낮아지고 있어서 그 실용적 기대치는 높아지고 있는 추세이다. 더욱이 최근에는 이러한 한계점을 극복하고 유전자칩 데이터로부터 유전자 조절네트워크를 추론해내는 다양한 연구들이 이루어지고 있다. 또 다른 예로는 세포내외부의 신호전달경로에 대한 수학모델을 정립하여 이로부터 예측되는 다양한 동역학 특성으로부터 기존에 설명되기 어려웠던 복잡한 현상들에 대한 새로운 통찰력을 얻고 이로부터 예측된 결과를 실험을 통해 검증해내는 연구로부터 암과 같은 질병의 발병기작과 전이특성 등을 새롭게 이해하려는 시도가 많은 주목을 받고 있다. 특히 동역학시스템의 비선형성은 직관적 예측과 전혀 다른 결과를 유발하곤 하는데 대부분의 세포내외동역학은 이러한 비선형 특성을 지니고 있어 시스템생물학적 접근 없이는 분석이나 예측이 불가능하다. 많은 실험생물학자들은 동일한 환경에서 배양한 세포들이 동일한 외부자극에 대해 서로 다르게 반응하는 현상을 의아해 하곤 하는데 이는 시스템의 상태변수개념을 간과하였기 때문에 겪는 일례라고 할 수 있다.
2000년대 이후로 국제학술대회와 각국의 연구그룹 및 연구기관 등에서 시스템생물학에 대한 중요도의 인식이 급격히 확산되고 있으며 관련 연구결과의 발표도 꾸준히 증가되고 있다. 다수의 국제저명학술지에서는 시스템생물학을 이미 주요 특집호로 다루었으며, 2004년에는 비로소 유럽 최대의 공학기관(IEE, 이후 IET로 변경되었음)으로부터 <시스템생물학(Systems Biology)>이라는 시스템생물학 분야 최초의 국제저널이 발간되기에 이르렀고(http://www.ieedl.org/IET-SYB) 이후 세계 각국의 여러 출판사들(Springer, Royal Academy of Science, Nature-EMBO 등)에서도 잇따른 관련국제저널들이 발간되어 이제는 학제간 융합연구영역을 넘어 독자적인 패러다임으로서 정립되기에 이르렀다. 뿐만 아니라 전세계의 유수한 대학에서는 시스템생물학을 새로운 학제분야로 받아들이고 이에 따른 학과를 신설하거나 연구프로그램을 가동하고 있는데 급기야는 보수적 아카데미의 대명사인 하버드대학에서도 ‘시스템생물학과’가 신설되기에 이르렀다. 이러한 교육, 연구프로그램들은 대부분의 경우 제어공학, 생명과학, 수학, 컴퓨터과학 등을 포함한 융합분야로 구성되어 있다. 시스템생물학은 최근 새로운 분야로 재등장하였음에도 불구하고 이미 개인수준의 인식을 넘어서 연구기관과 대학, 그리고 국가주도의 연구프로그램으로 확대되고 있다. 특히, 일본, 미국, 영국, 유럽의 여러 국가들을 위주로 연구비 투자와 저변확대가 경쟁적으로 이어지고 있으며 최근에는 중국에서도 국제학술회의 유치와 연구기관 설치에 막대한 투자를 전개하고 있다. 일본의 경우 동경에서 제1회 시스템생물학 국제학술대회를 개최한 데 이어 시스템생물학 연구소를 설립하고 2000년 초부터 이미 정부지원하의 대규모 국제프로젝트를 가동해오고 있으며, 미국에서도 비슷한 시점에 시스템생물학 연구소를 시애틀에 설립한 이래로 해마다 투자규모를 늘려 현재 미국 전역에 다수의 시스템생물학 연구센터와 연구컨소시엄이 구성 운영되고 있다. 또한 캐나다 오타와에도 시스템생물학 연구소가 설립 운영되고 있다. 독일에서는 2003년부터 대규모 시스템생물학 국가프로젝트를 가동하고 있고, 영국에서는 2006년 전국에 5개의 시스템생물학 핵심연구센터가 설립되었으며, 스웨덴, 프랑스, 이탈리아, 네덜란드, 이스라엘, 룩셈부르크, 아일랜드, 스위스, 덴마크, 호주 등 많은 유럽과 오세아니아 국가에서 이 분야에 앞다투어 투자를 기울이고 있다. 특히 아일랜드는 2004년 대규모 시스템생물학 연구프로젝트를 시작한 이후 2009년 더블린에 시스템생물학 연구소를 설립하며 이 분야에 막대한 투자를 주도하고 있다. 또한 유럽연합(EU)에서도 모든 유럽연합 국가들을 대상으로 하는 연구프로그램인 EU프레임워크-7에 시스템생물학을 주요 연구주제로 선정하여 여러 연구컨소시엄을 장기간 지원하고 있다. 현재 시스템생물학은 기초연구 차원을 넘어서서 이를 활용한 새로운 산업 창출로까지 이어지고 있다. 한편 국내에서도 2003년 서울 시스템생물학 국제심포지엄을 효시로 관심이 증대되고 있으며 Bio-Vision 2006 등 국가생명공학육성정책수립의 핵심에 시스템생물학을 언급하고 있다. 우리 연구실은 1999년 설립된 이래 전 세계적으로도 최초로 시스템생물학 연구를 독자 개척하여왔으며 시스템생물학의 기초로부터 응용에 이르는 다양한 연구를 선도해오고 있다. 우리 연구실의 이러한 독자적인 시도는 소위 ‘IT와 BT 융합연구’의 효시가 되었다. 대부분의 선진국에서 시스템생물학 분야에 집중적인 투자를 기울이고 있는 상황에 비하면 국내에서는 아직 그 중요도의 인식이나 투자규모 면에서 미미하지만 사회적으로 ‘융복합’ 연구가 주목을 받으면서 점차 인식이 확대되고 있다. 특히 국내의 고도화된 IT (인적, 물적) 인프라를 고려할 때 시스템생물학 융합연구는 앞으로 가파르게 성장할 것으로 기대되며 기초연구의 발전과 더불어 새로운 산업 창출로 이어질 것으로 예견된다.
많은 사람들이 20세기 초엽을 물리학의 전성시대라고 한다면 21세기는 생명과학의 전성시대가 될 것이라고 한다. 하지만 동시에 어떤 사람들은 정반대급부로 현재가 생명과학의 위기의 시대라고도 한다. 이는 생명현상의 이해가 어느 시점부터 그 복잡성으로 인해 정체되지 않고, 그러한 복잡한 생명현상을 새로운 시각에서 이해하며 이를 토대로 질병을 극복하기 위해서는 생명과학이 더 이상 실험자의 경험과 숙련된 기술에 크게 의존하는 ‘발견’의 학문이 아닌 동작원리를 설명할 수 있고 재현 가능한 ‘논리’의 학문으로서 거듭나야 한다는 것을 강조하는 의미이다. 즉, 복잡한 현상에 관여되는 중요한 구성요소들을 발견하는 차원에서 더 나아가 그러한 구성요소들이 어떠한 인과관계와 작동원리를 통해서 상호작용을 일으켜 비로소 관측된 복잡한 현상에 이르게 되는지를 논리적으로 설명할 수 있는 새로운 차원의 생명과학, 즉 시스템생물학으로 거듭날 때 생명현상의 수수께끼를 푸는데 한 발자국 더 가까이 다가갈 수 있음을 의미하는 것이다.
시스템생물학에 대한 사회의 기대는 단연 질병극복에 관한 것이다. 이는 지금까지 의생명과학의 접근이 주로 단일 유발인자(유전자, 단백질 등)로 부터 특정 질병의 발생을 선형적으로 연결하고자 하였던 시도의 실패와 반성에 기인하기도 한다. 인간유전체 프로젝트가 종료되고 첫 휴먼게놈지도의 초안이 발표된 후 10여년이 지난 시점에 <사이언스>지의 편집총론으로 게재된 “유전체만으론 부족하다(Genome is not enough)”란 기사에서, 유전체 서열 정보를 모두 알더라도 실제 많은 질병에 대한 우리의 이해는 달라질 것이 별로 없다는 충격적 메시지가 시사하는 바는 사실상 시스템생물학 관점에서 이미 예고된 것이었다. 이와 유사한 지적은 공학잡지인 <미국전기전자공학회 회로 및 시스템 매거진 (IEEE Circuits and Systems Magazine)>의 생명과학특집호(2012년)에서도 찾아볼 수 있다. 이 잡지의 기사에서는 가까운 미래에 많은 사람들이 기대하는 휴먼게놈 1천불이하의 시대가 도래하더라도 그렇게 얻어진 데이터로부터 유익한 정보를 찾기 위해서는 또다시 그 수만배에 달하는 비용을 치러야할 것이며, 어떤 신약이 통계적으로 환자 5%에게 효능이 있다는 것보다 과연 그 5%를 어떻게 구분해낼 것인가가 중요하다는 논의로부터 시스템생물학의 중요성을 강조하고 있다. 사실 단일 유전자의 발현 이상만으로 유발되는 멘델리안 유전질환은 극히 일부이며 대부분의 질병은 이와 같이 단일 유전자 또는 단백질의 기능 이상으로 설명하기 어려운, 소위 복잡계 질환이다. 중요한 것은 특정 분자가 아닌 다수 분자들의 조절네트워크에 문제의 원인이 있다는 것이다. 즉, 모든 유전자나 단백질들은 이러한 거대 네트워크의 한 구성요소일 뿐이며 어떤 특정 요소에 이상이 발생하더라도 대부분의 경우 네트워크의 적응조절기능에 의해 우회경로를 통하여 정상 동작이 가능하다. 따라서 생명현상의 조절과 질병발생은 단일 인자 차원이 아니라 여러 조절인자들의 복잡한 네트워크 관점에서 새롭게 조망되어야 한다. 이러한 관점에서 시스템생물학이 가까운 미래에 의생명과학 발전에 미칠 영향은 매우 지대할 것으로 보인다.
질병발생의 메커니즘에 대한 새로운 시스템 차원의 이해와 개인 맞춤형 치료라는 응용을 넘어서서, 생명에 관한 연구의 근저에는 언제나 생명의 기원과 진화의 원리에 대한 호기심이 남아있다. 시스템생물학 연구의 발전은 이러한 원천적 질문에도 새로운 발상의 대답을 제공하게 될 것이다. 이를 위하여 분자수준 또는 세포 그 이상의 수준에서 서로 상호작용하며 형성된 거대 네트워크의 어떠한 특정 구조가 ‘창발’하는 현상을 빚어내는지에 대한 심도 깊은 연구가 필요하다. 진화하는 네트워크의 구조는 과연 어느 순간 생명의 특징을 만들어내는 것일까?
시스템생물학의 미래는 일일이 열거하기 어려울 정도로 다양하며 유망하고 또한 복잡하다. 그러나 시스템생물학의 가장 큰 성과는 지금껏 많은 시행착오를 통한 우연적 발견의 과정으로 발전되어져 온 생명과학 연구에 커다란 피드백루프를 연결하는 것이라고 할 수 있다. 이 피드백루프는 어떠한 발견이 왜 이루어지게 되었는지, 또 예상과 다른 결과는 왜 관측이 된 것인지에 대한 ‘논리’를 제공해줌으로써 생명과학을 더 이상 우연한 발견의 학문이 아닌 논리적 접근과 분석이 가능한 영역으로 패러다임을 바꾸게 할 것이다. 시스템생물학은 호기심과 열정으로 가득한 과학자들의 융합학문이며, 지금 이 순간도 끊임없이 진화하고 있다.
최근 시스템생물학은 단일세포 오믹스 측정기술에 힘입어 새로운 르네상스를 맞이하고 있다. 특히 한꺼번에 단일세포수준에서 다양한 오믹스 데이터를 측정하고 이를 pseudo-time ordering을 통해 마치 time-series 측정을 한 것과 같은 시간에 따른 변화정보를 추출할 수 있게 됨으로써 과거에 수많은 노력을 통해서만 측정이 가능했던 세포내 변화에 대한 정보를 바탕으로 보다 정밀한 세포내 분자네트워크 모델을 만드는 것이 가능해졌으며 이로부터 세포운명 변화의 핵심메커니즘을 단일세포 수준에서 정밀하게 파악할 수 있게 되었고 나아가 이를 원하는 방향으로 제어하는 것이 가능해진 것이다. 이러한 단일세포 멀티오믹스 데이터 기반의 단일세포 시스템생물학은 생명의 근원에 대한 해답과 인류 난치질환의 새로운 치료기술에 대한 답을 제공해줄 것이다. 우리 연구실은 특히 암과 노화의 수수께끼를 풀고 이를 가역화할 수 있는 새로운 길을 찾고자 한다.
(참고문헌: 조광현, 시스템생물학, 홍릉과학출판사, 2013)